Van medicijnontwikkeling en voedselproductie tot recycling en gewasbescherming: in allerlei industrieën is er een revolutie op komst dankzij het computermodel AlphaFold, dat de vorm van eiwitstructuren voorspelt. Van dat model zijn inmiddels drie versies ontwikkeld, waarvan de tweede – AlphaFold2 – dit jaar zelfs in de prijzen is gevallen. Dinsdag wordt in Stockholm de Nobelprijs voor Scheikunde uitgereikt aan onder andere twee medewerkers van DeepMind (de AI-tak van Google) die het model ontwikkelden.
De hoofdrolspelers in dit verhaal
In elke cel van het lichaam worden eiwitten aangemaakt, die binnen en buiten de cel belangrijke rollen vervullen, bijvoorbeeld:
Elk eiwit bestaat uit een opgevouwen keten van aminozuren. Elke aminozuurketen vouwt automatisch op een unieke manier op, en dat patroon bepaalt de functie van het eiwit.
Je kunt het vergelijken met een kralenketting die altijd op dezelfde manier in de war raakt.
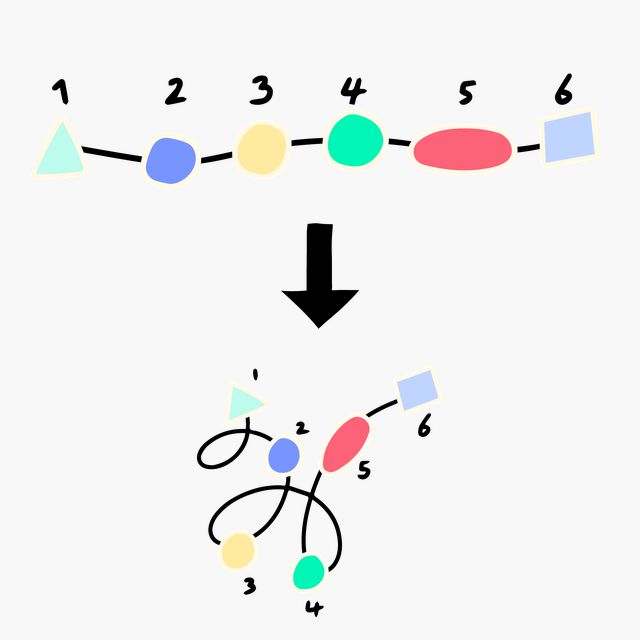
Sommige eiwitten behoren bijvoorbeeld tot de alfa-eiwitten: de aminozuurketen bestaat dan voor een belangrijk deel uit een rechtsdraaiende helix. Dat betekent dat de kralenketting is opgekruld tot een driedimensionale spiraal, die met de klok mee van je af draait.

Het probleem
Er zijn heel veel eiwitten waarvan de vorm en dus ook de functie nog onbekend zijn.
Voorheen kostte het maanden tijd – en veel geld – om een eiwitstructuur te bepalen, met een methode die kristallografie heet. Nu kan dat in minuten dankzij AlphaFold.
Hierdoor kunnen onderzoekers onder andere:
Ziektes beter en eerder begrijpen
(bij de ziekte van Alzheimer spelen
eiwitten een belangrijke rol).
Medicijnen ontwikkelen om die
ziektes te bestrijden.
Innovatieve eiwittoepassingen maken om bijvoorbeeld CO2 uit de atmosfeer te halen.
De oplossing
Google heeft een computerprogramma ontworpen: AlphaFold, een neuraal netwerk dat op basis van grote datasets van aminozuurketens driedimensionale eiwitstructuren beter en sneller kan voorspellen. Een neuraal netwerk is een vorm van kunstmatige intelligentie. Je kunt het zien als een kunstmatig model van het brein, dat je met digitale data traint.
Door die training kan het model patronen herkennen en een vermoedelijke uitkomst geven. Daar zijn al andere voorbeelden van. Bij ChatGPT is dat bijvoorbeeld het meest waarschijnlijke volgende woord in een zin en bij de app Obsidentify wordt op basis van een gemaakte natuurfoto bepaald wat de meest waarschijnlijke planten-, dieren- of paddenstoelensoort is.
Van AlphaFold zijn inmiddels drie versies ontwikkeld, waarvan AlphaFold2 recent de Nobelprijs voor Scheikunde heeft ontvangen.
Hoe werkt AlphaFold2?
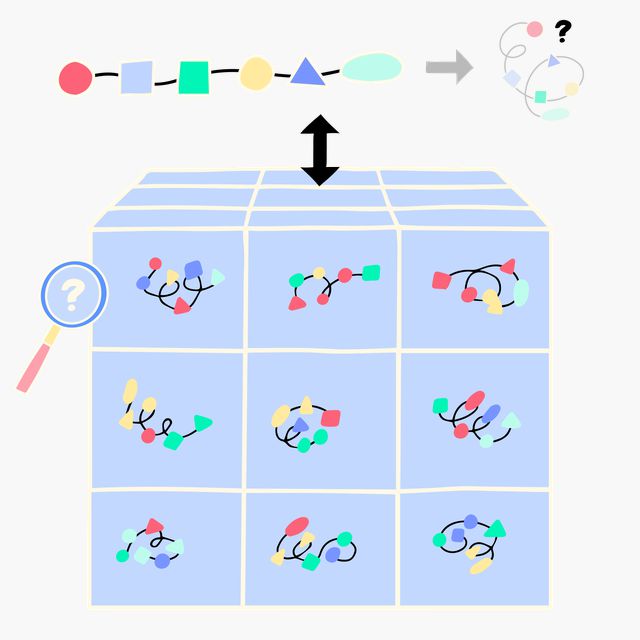
Het AlphaFold2-model is getraind met data van aminozuurketens waarvan de eiwitstructuren al bekend zijn. Hierdoor kan het model patronen herkennen in andere aminozuurketens (waarvan de eiwitstructuur nog niet bekend is) en zo de daarbij behorende eiwitstructuren beter en sneller voorspellen. Hoe dit werkt, leggen we hieronder stapsgewijs uit.
1.
Aan de basis van AlphaFold2 ligt een database van 200.000 bekende eiwitstructuren die eerder al zijn ontrafeld dankzij kristallografie.
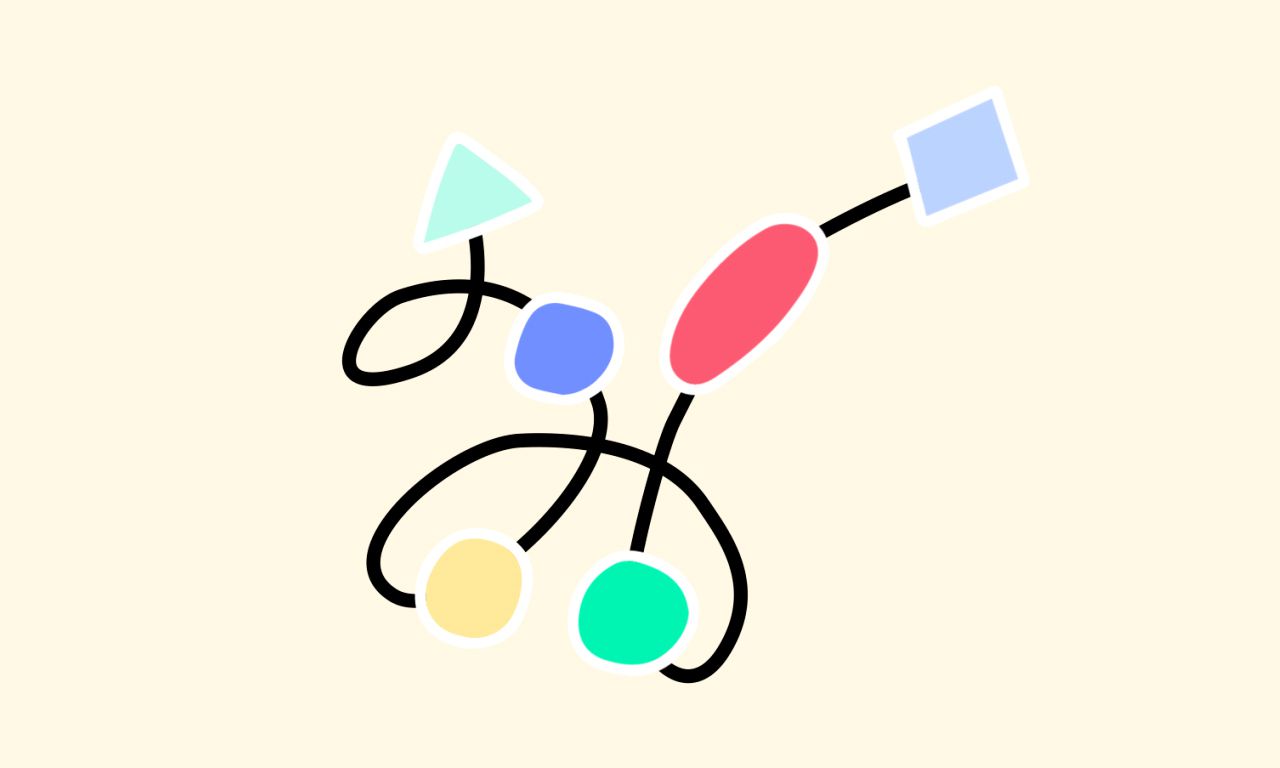
AlphaFold2 vergelijkt een aminozuurketen waarvan de vorm nog niet bekend is allereerst met aminzouurketens van eiwitten in die database.
2
Ook verkent het welke interacties de verschillende aminozuren met elkaar aangaan:
3
Dan komt het model met een inschatting hoe stukjes van de aminozuurketen zich in de ruimte tot elkaar kunnen verhouden. Je kunt het zien als de kralenketting die op een bepaalde manier in een 3D-ruimte wordt geplaatst.
4
Vervolgens wordt op dit 3D-model een vorm van kunstmatige intelligentie toegepast: een specifiek neuraal netwerk, dat de ‘Transformer’ heet. Je kunt dit zien als een machine waarin het 3D-model wordt geladen. Die machine kan dan heel snel bepalen welke stukjes informatie van belang zijn, en dus kunnen bijdragen aan een relevante uitkomst.
AlphaFold3
In mei dit jaar verscheen AlphaFold3, dat met nog geavanceerdere technieken nóg nauwkeurigere voorspellingen kan maken. Ook de structuren van dna en rna zijn ermee na te bootsen. Of zoals de ontwikkelaars van AlphaFold3 het zelf omschreven bij de lancering in mei: ‘hiermee kunnen we de structuur en interacties van alle moleculen van levende wezens voorspellen, met ongeëvenaarde precisie’.
In totaal zijn er in de open database nu meer dan 200 miljoen voorspelde structuren van eiwitten beschikbaar.
Waarvoor wordt AlphaFold al gebruikt?



/s3/static.nrc.nl/images/gn4/stripped/data134707599-d00030.jpg|https://images.nrc.nl/vpnUM9Q_Xyiu79vZOXD4XJgvcUw=/1920x/filters:no_upscale()/s3/static.nrc.nl/images/gn4/stripped/data134707599-d00030.jpg|https://images.nrc.nl/5rbSD4xqDO6K-FYQLgabwoiwtWY=/5760x/filters:no_upscale()/s3/static.nrc.nl/images/gn4/stripped/data134707599-d00030.jpg)

/s3/static.nrc.nl/images/gn4/stripped/data134707695-1aa407.jpg|https://images.nrc.nl/f2Fl3ts7H3chNrdocE5deTBHtRA=/1920x/filters:no_upscale()/s3/static.nrc.nl/images/gn4/stripped/data134707695-1aa407.jpg|https://images.nrc.nl/-HcVpkITZ8SF5zpaos26uxvoXzw=/5760x/filters:no_upscale()/s3/static.nrc.nl/images/gn4/stripped/data134707695-1aa407.jpg)
/s3/static.nrc.nl/images/gn4/stripped/data134707527-9b78f0.jpg|https://images.nrc.nl/egHoArt1hjT4jJ8WiblaXhtbWS4=/1920x/filters:no_upscale()/s3/static.nrc.nl/images/gn4/stripped/data134707527-9b78f0.jpg|https://images.nrc.nl/GrOOxAKkbmpJ1ibPH49M03Bbh9w=/5760x/filters:no_upscale()/s3/static.nrc.nl/images/gn4/stripped/data134707527-9b78f0.jpg)
/s3/static.nrc.nl/images/gn4/stripped/data134707752-336633.jpg|https://images.nrc.nl/gGCDGouxSp__yT0ujUsc_h7fE20=/1920x/filters:no_upscale()/s3/static.nrc.nl/images/gn4/stripped/data134707752-336633.jpg|https://images.nrc.nl/rkSbvw4jFG04G4bUuF4wfLFkSgs=/5760x/filters:no_upscale()/s3/static.nrc.nl/images/gn4/stripped/data134707752-336633.jpg)



